
10 Tidyverse Funktionen, die den Tag retten können
In diesem Beitrag stellen wir 10 Tidyverse Funktionen vor, die von Beginnern häufig übersehen werden, sich im passenden Kontext jedoch als sehr nützlich erweisen. Dabei schildern wir zunächst ein Problem, welches uns in ähnlicher Form in der Praxis begegnet und erklären anschließend, wie uns das Tidyverse dabei hilft, dieses Problem zu lösen.

10 Tidyverse Funktionen, die den Tag retten können
In diesem Beitrag stellen wir 10 Tidyverse Funktionen vor, die von Beginnern häufig übersehen werden, sich im passenden Kontext jedoch als sehr nützlich erweisen. Dabei schildern wir stets zunächst ein Problem, welches uns in ähnlicher Form in der Praxis begegnet und erklären anschließend, wie uns das Tidyverse dabei hilft, dieses Problem zu lösen.
Für die Aufbereitung und Analyse von Daten in R haben sich die Tidyverse Pakete in den letzten Jahren als Branchenstandard etabliert. Wir bei eoda nutzen viele Funktionen aus dem Tidyverse, um unsere tägliche Arbeit effizienter zu gestalten.
Inhalt
- crossing
- rowwise
- pluck
- rownames_to_column & rowid_to_column
- parse_number
- fct_lump_*
- fct_reorder + geom_col
- separate & separate_rows
- str_flatten_comma
- arrange + distinct
- Fazit
Wir beginnen erwartungsgemäß mit dem Laden der notwendigen Bibliotheken:
library(tidyverse)1. crossing
Problem:
Für das erste Beispiel betrachten wir eine statistische Anwendung. Ausgehend von zwei Vektoren numerischer Mittelwerte und Standardabweichungen sollen alle Kombination der auftretenden Werte in einem Data Frame gesammelt werden.
Lösung:
Die crossing() Funktion aus dem tidyr package erfüllt genau diesen Zweck. Sie nimmt eine beliebige Anzahl an Vektoren als Input und bildet alle möglichen Kombinationen der auftretenden Werte:
means <- c(-1, 0, 1)
standard_deviations <- c(0.1, 0.5, 1)
mean_sd_combinations <- crossing(means, standard_deviations)
mean_sd_combinationsCode-Sprache: R (r)| means | standard_deviations |
|---|---|
| -1 | 0.1 |
| -1 | 0.5 |
| -1 | 1.0 |
| 0 | 0.1 |
| 0 | 0.5 |
| 0 | 1.0 |
| 1 | 0.1 |
| 1 | 0.5 |
| 1 | 1.0 |
Bonus:
crossing() kann nicht nur Vektoren, sondern auch Data Frames als Input nehmen. In diesem Fall werden alle Kombinationen der Zeilen gebildet.
Das ist besonders nützlich, wenn eines der Data Frames “globale” Informationen bereit stellt (im folgenden Beispiel population_data), welche für alle Beobachtungen gültig sind und das zweite Data Frame “lokale” Informationen, die sich zwischen Beobachtungen oder Gruppen unterscheiden (im Beispiel group_data).
population_data <- tibble(
global_feature_1 = "e",
global_feature_2 = 5,
)
population_dataCode-Sprache: R (r)| global_feature_1 | global_feature_2 |
|---|---|
| e | 5 |
group_data <- tibble(
group = 1:3,
local_feature_1 = c(2, 5, 3),
local_feature_2 = c(TRUE, FALSE, FALSE)
)
group_dataCode-Sprache: R (r)| group | local_feature_1 | local_feature_2 |
|---|---|---|
| 1 | 2 | TRUE |
| 2 | 5 | FALSE |
| 3 | 3 | FALSE |
Als Ergebnis von crossing() erhalten wir ein einziges Data Frame, in dem jede Zeile sowohl die globalen als auch die gruppenspezifischen Werte enthält:
crossing(population_data, group_data)| global_feature_1 | global_feature_2 | group | local_feature_1 | local_feature_2 |
|---|---|---|---|---|
| e | 5 | 1 | 2 | TRUE |
| e | 5 | 2 | 5 | FALSE |
| e | 5 | 3 | 3 | FALSE |
2. rowwise
Problem:
Wir bleiben bei dem Anwendungsbeispiel aus Abschnitt 1. Für jede der Mittelwert-Standardabweichung Kombinationen sollen fünf zufällige Werte (samples) einer Standardnormalverteilung gezogen und dem Data Frame in einer neuen Spalte hinzugefügt werden.
Folglich müssen wir hier auf Zeilenebene agieren: Jede Zeile des Data Frames bildet eine zusammengehörende Einheit. Die neu generierten Werte der ersten Zeile basieren ausschließlich auf den restlichen Werten der ersten Zeile.
Eine weitere Besonderheit ist, dass wir nicht nur einen einzelnen, sondern mehrere Einträge pro Zelle hinzufügen. Damit dies mit der Struktur eines Data Frames kompatibel ist, müssen diese in einer Liste zusammengefasst werden. Die neue Spalte ist folglich eine list column – eine Spalte bestehend aus Listen.
Lösung:
Eine Möglichkeit besteht in der Nutzung der map() Familie aus dem purrr Paket. Die Spalten means und standard_deviations, auf welche die rnorm() Funktion angewandt wird, werden durch die Platzhalter .x und .y referenziert:
random_samples_map <- mean_sd_combinations |> mutate(
samples = map2(means, standard_deviations, ~ rnorm(n = 5, mean = .x, sd = .y))
)
random_samples_map |> head()
<em>## # A tibble: 6 × 3</em>
<em>## means standard_deviations samples </em>
<em>## <dbl> <dbl> <list> </em>
<em>## 1 -1 0.1 <dbl [5]></em>
<em>## 2 -1 0.5 <dbl [5]></em>
<em>## 3 -1 1 <dbl [5]></em>
<em>## 4 0 0.1 <dbl [5]></em>
<em>## 5 0 0.5 <dbl [5]></em>
<em>## 6 0 1 <dbl [5]></em>Code-Sprache: R (r)Jeder Eintrag der neuen samples Spalte besteht aus einer Liste mit fünf gezogenen Werten aus einer Standardnormalverteilung:
random_samples_map$samples[[1]]
<em>## [1] -1.0416796 -0.9907691 -0.9249944 -0.8859866 -1.0676741</em>Code-Sprache: R (r)Für viele Anwendungsfälle bietet die rowwise() Funktion aus dem dplyr Paket eine anwendungsfreundlichere Alternative. Die Spaltennamen means und standard_deviations können hierbei direkt im Aufruf der rnorm() Funktion ohne die Verwendung von Platzhaltern genutzt werden.
Da die neue Spalte aus Listen besteht, muss der Aufruf von rnorm() innerhalb von list() erfolgen:
random_samples_map <- mean_sd_combinations |>
dplyr::rowwise() |>
mutate(samples = list(rnorm(n = 5, mean = means, sd = standard_deviations)))
random_samples_map$samples[[1]]
<em>## [1] -0.9437694 -0.9311953 -1.0259749 -1.0115392 -1.0614477</em>Code-Sprache: R (r)Bonus:
Bei der Arbeit mit ‘list columns’ kann die dplyr Funktion nest_by() sehr nützlich sein, welche anders als tidyr::nest() zeilenweise Gruppen bildet.
Als Beispiel bilden wir eine eigene Gruppe für jeden cyl (cylinder) Wert aus dem mtcars Datensatz. Alle verbleibenden mtcars Spalten werden in einer neuen Spalte bestehend aus Data Frames gebündelt.
mtcars |> nest_by(cyl)
<em>## # A tibble: 3 × 2</em>
<em>## # Rowwise: cyl</em>
<em>## cyl data</em>
<em>## <dbl> <list<tibble[,10]>></em>
<em>## 1 4 [11 × 10]</em>
<em>## 2 6 [7 × 10]</em>
<em>## 3 8 [14 × 10]</em>Code-Sprache: R (r)Hiervon ausgehend können wir eine neue Spalte mit linearen Modellen von mpg (miles per gallon) in Abhängigkeit von hp (horse power) hinzufügen.
In einem letzten Schritt extrahieren wir aus diesem die Steigungskoeffizienten, also je eine Zahl pro Zylinderwert. Das Ergebnis ist ein einziges Data Frame, in dem die ursprünglichen Daten, die Modellobjekte und die Steigungskoeffzienten enthalten sind:
mtcars |>
nest_by(cyl) |>
mutate(model = list(lm(mpg ~ hp, data = data))) |>
mutate(slope = coef(model)[2])
<em>## # A tibble: 3 × 4</em>
<em>## # Rowwise: cyl</em>
<em>## cyl data model slope</em>
<em>## <dbl> <list<tibble[,10]>> <list> <dbl></em>
<em>## 1 4 [11 × 10] <lm> -0.113 </em>
<em>## 2 6 [7 × 10] <lm> -0.00761</em>
<em>## 3 8 [14 × 10] <lm> -0.0142</em>Code-Sprache: R (r)3. pluck
Problem:
Aus der genesteten Liste l soll der String "c" der untersten Ebene ausgewählt werden, d.h. der dritte Wert des Elements b im ersten Listenelement von a. Insgesamt müssen wir also einen Wert aus dem vierten Level der Liste extrahieren.
l <- list(a = list(c(1, 2, list(b = c("a", "b", "c")))))
l
<em>## $a</em>
<em>## $a[[1]]</em>
<em>## $a[[1]][[1]]</em>
<em>## [1] 1</em>
<em>## </em>
<em>## $a[[1]][[2]]</em>
<em>## [1] 2</em>
<em>## </em>
<em>## $a[[1]]$b</em>
<em>## [1] "a" "b" "c"</em>Code-Sprache: R (r)Lösung:
Dies ist natürlich auch ohne zusätzliche Pakete möglich, jedoch vergleichsweise schwierig zu lesen:
l$a[[1]]$b[3]
<em>## [1] "c"</em>Code-Sprache: R (r)pluck() aus dem purrr Package löst die Aufgabe hingegen sehr elegant und leicht verständlich. Der Name bzw. Index jedes Levels der Liste wird einfach sequentiell als Argument der Funktion übergeben:
l |> purrr::pluck("a", 1, "b", 3)
<em>## [1] "c"</em>Code-Sprache: R (r)4. rownames_to_column & rowid_to_column
Problem 1:
Die Zeilennamen eines Datensatzes sollen in die erste Spalte geschrieben werden. Als Beispiel wählen wir den bekannten mtcars Datensatz. In diesem beschreiben die Zeilennamen das Modell des Autos, welche einer neuen model Spalte hinzugefügt werden sollen:
mtcars |> head()| mpg | cyl | disp | hp | drat | wt | qsec | vs | am | gear | carb | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Mazda RX4 | 21.0 | 6 | 160 | 110 | 3.90 | 2.620 | 16.46 | 0 | 1 | 4 | 4 |
| Mazda RX4 Wag | 21.0 | 6 | 160 | 110 | 3.90 | 2.875 | 17.02 | 0 | 1 | 4 | 4 |
| Datsun 710 | 22.8 | 4 | 108 | 93 | 3.85 | 2.320 | 18.61 | 1 | 1 | 4 | 1 |
| Hornet 4 Drive | 21.4 | 6 | 258 | 110 | 3.08 | 3.215 | 19.44 | 1 | 0 | 3 | 1 |
| Hornet Sportabout | 18.7 | 8 | 360 | 175 | 3.15 | 3.440 | 17.02 | 0 | 0 | 3 | 2 |
| Valiant | 18.1 | 6 | 225 | 105 | 2.76 | 3.460 | 20.22 | 1 | 0 | 3 | 1 |
Lösung:
Das tibble Paket stellt die Funktion rownames_to_column() bereit. Dem Parameter var kann dabei ein String mit dem gewünschten neuen Spaltennamen übergeben werden. Die neue Spalte wird automatisch an die erste Position des Datensatzes gesetzt.
mtcars_model <- mtcars |> tibble::rownames_to_column(var = "model")
mtcars_model |> head()Code-Sprache: R (r)| model | mpg | cyl | disp | hp | drat | wt | qsec | vs | am | gear | carb |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Mazda RX4 | 21.0 | 6 | 160 | 110 | 3.90 | 2.620 | 16.46 | 0 | 1 | 4 | 4 |
| Mazda RX4 Wag | 21.0 | 6 | 160 | 110 | 3.90 | 2.875 | 17.02 | 0 | 1 | 4 | 4 |
| Datsun 710 | 22.8 | 4 | 108 | 93 | 3.85 | 2.320 | 18.61 | 1 | 1 | 4 | 1 |
| Hornet 4 Drive | 21.4 | 6 | 258 | 110 | 3.08 | 3.215 | 19.44 | 1 | 0 | 3 | 1 |
| Hornet Sportabout | 18.7 | 8 | 360 | 175 | 3.15 | 3.440 | 17.02 | 0 | 0 | 3 | 2 |
| Valiant | 18.1 | 6 | 225 | 105 | 2.76 | 3.460 | 20.22 | 1 | 0 | 3 | 1 |
Problem 2:
Im zweiten Schritt soll eine index Spalte hinzugefügt werden, die jede Beobachtung eindeutig durch eine ID identifiziert. Dafür nummerieren wir einfach die Zeilen durch und schreiben die Zeilennummern in die neue Spalte.
Lösung:
Eine naheliegende Lösung erstellt mittels mutate() in Kombination mit nrow() oder dplyr::row_number() eine neue Spalte und setzt diese mit relocate() an die erste Position:
mtcars_model |>
<em># alternativ: mutate(index = row_number()) |></em>
mutate(index = 1:nrow(mtcars)) |>
relocate(index) |>
head()Code-Sprache: R (r)| index | model | mpg | cyl | disp | hp | drat | wt | qsec | vs | am | gear | carb |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | Mazda RX4 | 21.0 | 6 | 160 | 110 | 3.90 | 2.620 | 16.46 | 0 | 1 | 4 | 4 |
| 2 | Mazda RX4 Wag | 21.0 | 6 | 160 | 110 | 3.90 | 2.875 | 17.02 | 0 | 1 | 4 | 4 |
| 3 | Datsun 710 | 22.8 | 4 | 108 | 93 | 3.85 | 2.320 | 18.61 | 1 | 1 | 4 | 1 |
| 4 | Hornet 4 Drive | 21.4 | 6 | 258 | 110 | 3.08 | 3.215 | 19.44 | 1 | 0 | 3 | 1 |
| 5 | Hornet Sportabout | 18.7 | 8 | 360 | 175 | 3.15 | 3.440 | 17.02 | 0 | 0 | 3 | 2 |
| 6 | Valiant | 18.1 | 6 | 225 | 105 | 2.76 | 3.460 | 20.22 | 1 | 0 | 3 | 1 |
Erneut liefert das tibble Paket eine kompaktere Lösung. rowid_to_column() erledigt unsere Aufgabe in einem Schritt. Genau wie zuvor kann durch das var Argument der Name der neuen Spalte bestimmt werden:
mtcars_model |>
tibble::rowid_to_column(var = "index") |>
head()Code-Sprache: R (r)| index | model | mpg | cyl | disp | hp | drat | wt | qsec | vs | am | gear | carb |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | Mazda RX4 | 21.0 | 6 | 160 | 110 | 3.90 | 2.620 | 16.46 | 0 | 1 | 4 | 4 |
| 2 | Mazda RX4 Wag | 21.0 | 6 | 160 | 110 | 3.90 | 2.875 | 17.02 | 0 | 1 | 4 | 4 |
| 3 | Datsun 710 | 22.8 | 4 | 108 | 93 | 3.85 | 2.320 | 18.61 | 1 | 1 | 4 | 1 |
| 4 | Hornet 4 Drive | 21.4 | 6 | 258 | 110 | 3.08 | 3.215 | 19.44 | 1 | 0 | 3 | 1 |
| 5 | Hornet Sportabout | 18.7 | 8 | 360 | 175 | 3.15 | 3.440 | 17.02 | 0 | 0 | 3 | 2 |
| 6 | Valiant | 18.1 | 6 | 225 | 105 | 2.76 | 3.460 | 20.22 | 1 | 0 | 3 | 1 |
5. parse_number
Problem:
Bei der täglichen Arbeit mit Daten begegnen wir häufig Datensätzen, die vor der Weiterverwendung noch bereinigt werden müssen.
Der nachfolgende Datensatz enthält eine Spalte mit Produkten und eine weitere Spalte mit zugehörigen Preisen. Die Preise sind hierbei jedoch in einen String ohne feste Struktur eingebunden:
data_prices <- tibble(
product = 1:3,
costs = c("$10 -> expensive", "cheap: $2.50", "free, $0 !!")
)
data_pricesCode-Sprache: R (r)| product | costs |
|---|---|
| 1 | $10 -> expensive |
| 2 | cheap: $2.50 |
| 3 | free, $0 !! |
Die Aufgabe besteht nun darin, aus den Strings die numerischen Preises für jedes Produkt zu separieren.
Lösung:
Eine funktionierende, aber häufig umständliche Lösung besteht in der Nutzung regulärer Ausdrücke. In diesem Beispiel suchen wir nach dem ersten Match mindestens einer Ziffer gefolgt von optionalem Punkt und Nachkommastellen. Ein Nachteil dieses Ansatzes ist, dass die Ergebnisspalte immer noch vom Typ character ist:
one_or_more_digits <- "\\d+"
optional_dot <- "\\.?"
optional_digits <- "\\d*"
data_prices |> mutate(price = stringr::str_extract(
string = costs,
pattern = paste0(one_or_more_digits, optional_dot, optional_digits)
))
<em>## # A tibble: 3 × 3</em>
<em>## product costs price</em>
<em>## <int> <chr> <chr></em>
<em>## 1 1 $10 -> expensive 10 </em>
<em>## 2 2 cheap: $2.50 2.50 </em>
<em>## 3 3 free, $0 !! 0</em>Code-Sprache: R (r)Es gibt jedoch einen komfortableren Weg: Das readr package, welches üblicherweise zum Datenimport verwendet wird, stellt die Hilfsfunktion parse_number() bereit. Diese scannt einen Vektor mit Strings nach der ersten Zahl und extrahiert diese aus ihrem Kontext. Mögliche Nachkommastellen werden automatisch berücksichtigt.
Die neue price Spalte gehört in diesem Fall direkt dem Datentyp double an:
data_prices |> mutate(price = parse_number(costs))Code-Sprache: R (r)| product | costs | price |
|---|---|---|
| 1 | $10 -> expensive | 10.0 |
| 2 | cheap: $2.50 | 2.5 |
| 3 | free, $0 !! | 0.0 |
6. fct_lump_*
Problem:
In diesem Beispiel arbeiten wir mit dem babynames Datensatz aus dem gleichnamigen R Paket, welches die beliebtesten Babynamen in den USA über mehrere Jahrzehnte auflistet. Die Spalte n gibt die absolute Häufigkeit des Namens innerhalb eines Jahres an:
babynames::babynames |> head()Code-Sprache: R (r)| year | sex | name | n | prop |
|---|---|---|---|---|
| 1880 | F | Mary | 7065 | 0.0723836 |
| 1880 | F | Anna | 2604 | 0.0266790 |
| 1880 | F | Emma | 2003 | 0.0205215 |
| 1880 | F | Elizabeth | 1939 | 0.0198658 |
| 1880 | F | Minnie | 1746 | 0.0178884 |
| 1880 | F | Margaret | 1578 | 0.0161672 |
Wir interessieren uns dafür, auf welche Buchstaben Mädchennamen im Jahr 2000 am häufigsten enden:
names_2000 <- babynames::babynames |> filter(year == 2000)
last_letters_females <- names_2000 |>
mutate(last_letter = stringr::str_sub(name, start = -1, end = -1)) |>
filter(sex == "F") |>
count(last_letter, wt = n, name = "num_babies", sort = TRUE)
last_letters_females |> head(10)Code-Sprache: R (r)| last_letter | num_babies |
|---|---|
| a | 675963 |
| e | 318399 |
| n | 248450 |
| y | 246324 |
| h | 117324 |
| l | 56623 |
| r | 50769 |
| i | 42591 |
| s | 32603 |
| t | 9796 |
Einige Buchstaben stehen erwartungsgemäß wesentlich häufiger an letzter Stelle als andere. Zu Übersichtszwecken sollen alle Buchstaben mit geringer Häufigkeit zu einer gemeinsamen Other Kategorie zusammengefasst werden.
Lösung:
Das forcats Paket hilft uns hierbei. Die fct_lump_*() Familie aggregiert seltenere Werte einer Faktor- (oder hier Character-) Variablen nach verschiedenen Kriterien:
fct_lump_n()behält dienhäufigsten Werte und fasst alle anderen Werte zu einer neuen Kategorie zusammen.fct_lump_min()fasst alle Werte zusammen, welche seltener als eine gegebene absolute Häufigkeit vorkommen.fct_lump_prop()fasst alle Werte zusammen, welche seltener als eine gegebene relative Häufigkeit (Anteil zwischen 0 und 1) auftreten.fct_lump_lowfreq()fasst automatisch die seltensten Werte zusammen, sodass die aggregierteOtherKategorie immer noch die geringste Häufigkeit unter den neuen Kategorien besitzt.
In unserem Beispiel nutzen wir fct_lump_n() und behalten die häufigsten fünf letzten Buchstaben bei:
last_letters_females_lumped <- last_letters_females |>
mutate(last_letter = factor(last_letter) |> fct_lump_n(
n = 5, w = num_babies, other_level = "Other"
)) |>
count(
last_letter,
wt = num_babies, name = "num_babies", sort = TRUE
)
last_letters_females_lumpedCode-Sprache: R (r)| last_letter | num_babies |
|---|---|
| a | 675963 |
| e | 318399 |
| n | 248450 |
| y | 246324 |
| Other | 208650 |
| h | 117324 |
Der Parameter w (für weight) kann dabei optional eine Spalte angeben, deren Werte zur Bestimmung der Häufigkeit aufsummiert werden. Dies ist dann nützlich, wenn wie im obigen Beispiel jeder Buchstabe nur in einer Zeile vorkommt und die zugehörigen Häufigkeiten bereits berechnet wurden. Der Parameter wird nicht benötigt, falls die Häufigkeiten noch nicht berechnet wurden und jeder Buchstabe n Mal in der last_letter Spalte dupliziert wäre.
7. fct_reorder + geom_col
Problem:
Wir bleiben auch für dieses Beispiel bei dem babynames Datensatz und visualisieren die Anzahl der sechs häufigsten Mädchennamen in einem Balkendiagramm mit geom_col():
plot_color <- "#8bac37"
top_names_females <- names_2000 |>
filter(sex == "F") |>
slice_max(n, n = 6)
top_names_females |>
ggplot(aes(n, name)) +
geom_col(fill = plot_color) +
labs(
title = "Die 6 häufigsten Babynamen für Mädchen im Jahr 2000",
x = "Häufigkeit", y = NULL,
) +
theme_light() +
theme(plot.title = element_text(hjust = 0.5))Code-Sprache: R (r)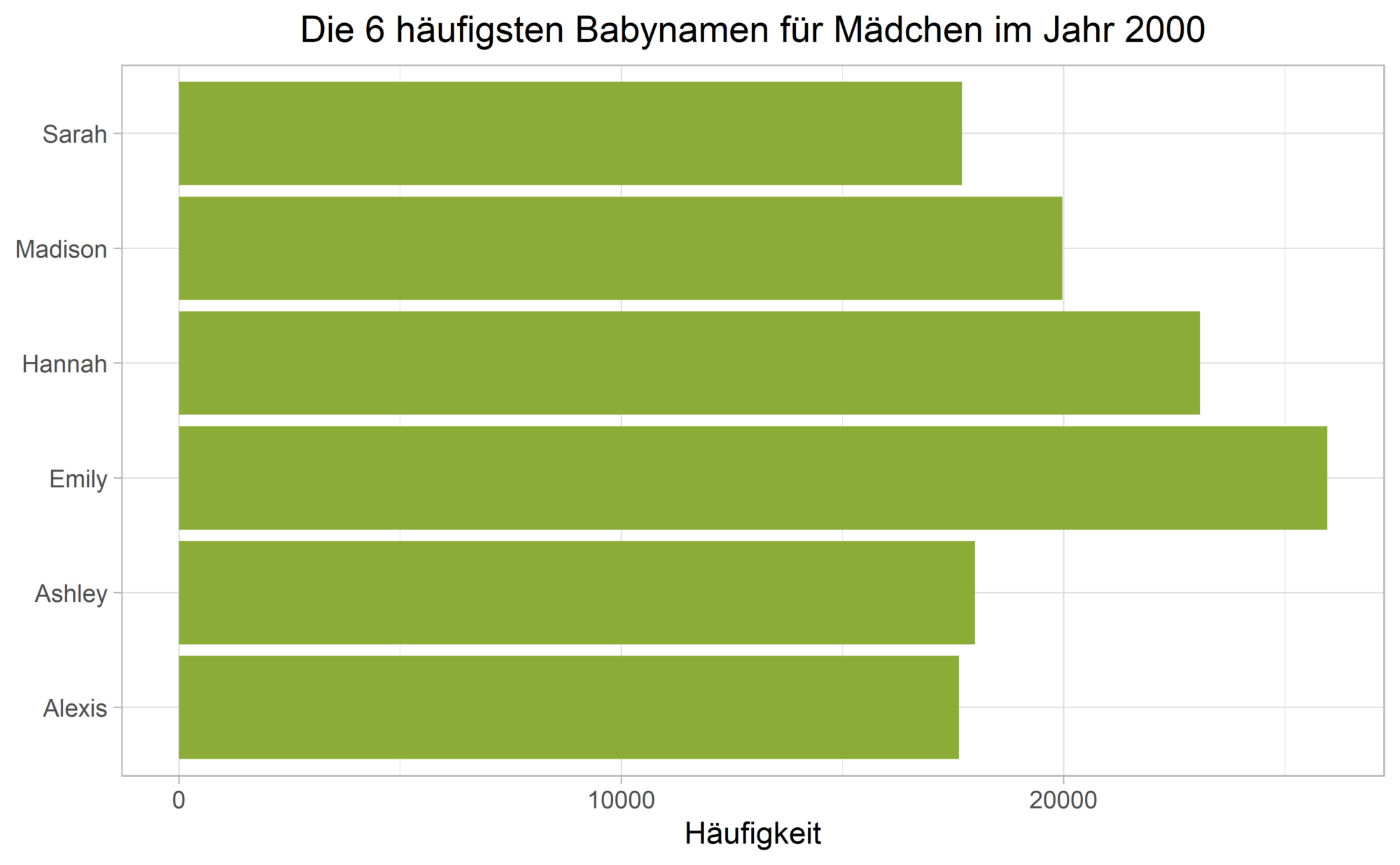
Die Namen sind entlang der y-Achse nicht nach ihrer Häufigkeit geordnet!
Lösung:
Um dies zu erreichen, sortieren wir die name Spalte gemäß ihrer Häufigkeit (der Spalte n) neu.
Dieser Fall tritt in der Praxis so häufig auf, dass ich geom_col() fast ausschließlich in Kombination mit fct_reorder() aus dem forcats Paket verwende:
top_names_females |>
mutate(name = fct_reorder(name, n)) |>
ggplot(aes(n, name)) +
geom_col(fill = plot_color) +
labs(
title = "Die 6 häufigsten Babynamen für Mädchen im Jahr 2000",
x = "Häufigkeit", y = NULL,
) +
theme_light() +
theme(plot.title = element_text(hjust = 0.5))Code-Sprache: R (r)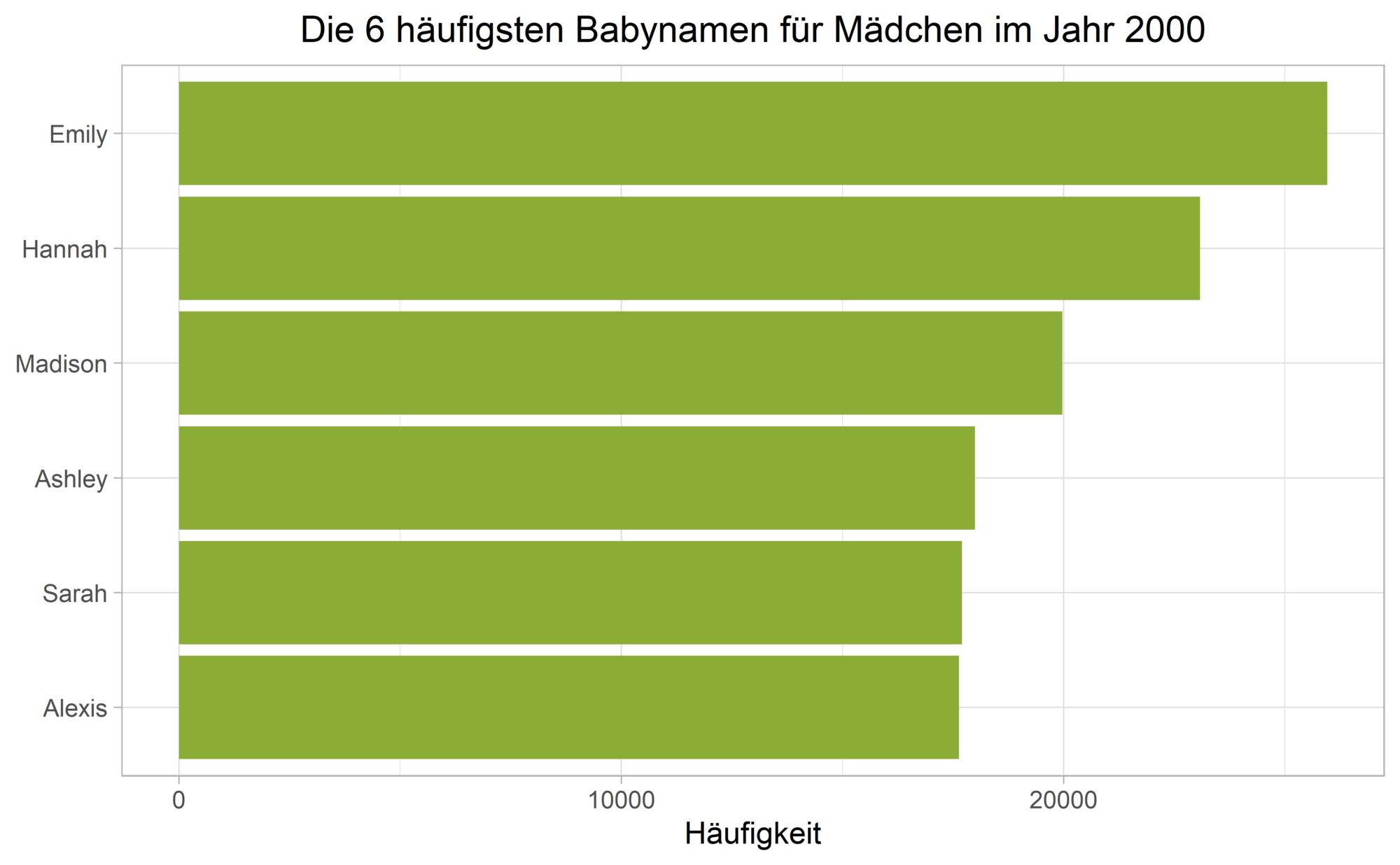
Bonus:
Das obige Vorgehen funktioniert nicht mehr so einfach, wenn für jeden Wert einer weiteren Faktorvariablen ein eigenes Balkendiagramm in absteigender Häufigkeit geplottet werden soll. Als Beispiel betrachten wir nun zusätzlich die häufigsten Jungennamen:
top_names <- names_2000 |>
group_by(sex) |>
slice_max(n, n = 6)Code-Sprache: R (r)Mit fct_reorder() werden die Balken in jedem Subplot stets gemäß ihrer Häufigkeit im gesamten Datensatz (und nicht nur innerhalb jedes Wertes der sex Variable) angeordnet.
Das tidytext Paket, welches primär zur Analyse von Textdaten verwendet wird, rettet uns an dieser Stelle.
Die Hilfsfunktionen reorder_within() und scale_y_reordered() erfüllen exakt den gewünschten Zweck und sortieren die Werte der Faktorvariablen innerhalb jedes Subplots:
top_names |>
mutate(name = tidytext::reorder_within(name, by = n, within = sex)) |>
ggplot(aes(n, name)) +
geom_col(fill = plot_color) +
labs(
title = "Die 6 häufigsten Babynamen für Mädchen und Jungs im Jahr 2000",
x = "Häufigkeit", y = NULL,
) +
facet_wrap(facets = vars(sex), scales = "free_y") +
tidytext::scale_y_reordered() +
theme_light() +
theme(plot.title = element_text(hjust = 0.5))Code-Sprache: R (r)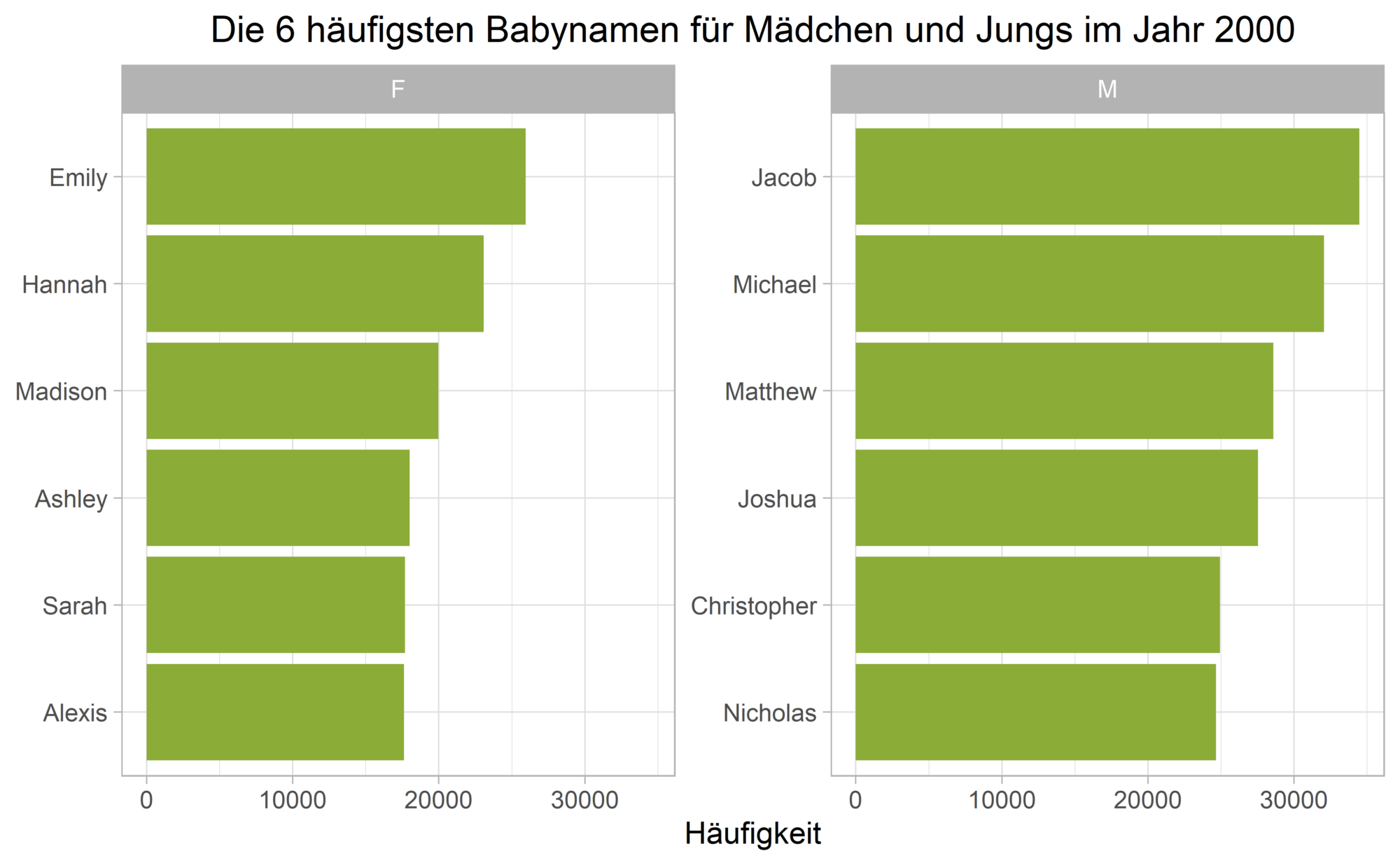
8. separate & separate_rows
Problem 1: Der folgende Datensatz soll die Ergebnisse verschiedener Länderspiele abbilden:
data_games <- tibble(
country = c("Germany", "France", "Spain"),
game = c("England - win", "Brazil - loss", "Portugal - tie")
)
data_gamesCode-Sprache: R (r)| country | game |
|---|---|
| Germany | England – win |
| France | Brazil – loss |
| Spain | Portugal – tie |
Die game Spalte umfasst jedoch zwei unterschiedliche Arten von Informationen: den Gegner sowie das Ergebnis.
Lösung:
Um das Data Frame tidy zu machen, splitten wir die game Spalte mit der separate() Funktion aus dem tidyr Paket in zwei Spalten auf:
data_games |> separate(col = game, into = c("opponent", "result"))Code-Sprache: R (r)| country | opponent | result |
|---|---|---|
| Germany | England | win |
| France | Brazil | loss |
| Spain | Portugal | tie |
Problem 2:
Ein ähnliches Problem tritt auf, wenn eine Spalte in jeder Zeile zwei Informationen des gleichen Typs enthält. Die opponent Spalte umfasst nun ausschließlich gegnerische Teams, jedoch gleich mehrere pro Zeile:
data_opponents <- tibble(
country = c("Germany", "France", "Spain"),
opponent = c("England, Switzerland", "Brazil, Denmark", "Portugal, Argentina")
)
data_opponentsCode-Sprache: R (r)| country | opponent |
|---|---|
| Germany | England, Switzerland |
| France | Brazil, Denmark |
| Spain | Portugal, Argentina |
Der gewünschte Output enthält in diesem Fall nicht mehr Spalten, sondern mehr Zeilen, je eine pro Gegner.
Lösung:
separate_rows() splittet jede Zeile der opponent Spalte in mehrere Zeilen, die korrespondierenden country Werte werden entsprechend dupliziert:
data_opponents |> separate_rows(opponent)Code-Sprache: R (r)| country | opponent |
|---|---|
| Germany | England |
| Germany | Switzerland |
| France | Brazil |
| France | Denmark |
| Spain | Portugal |
| Spain | Argentina |
9. str_flatten_comma
Problem:
Ein Vektor aus Strings soll zu einem einzigen String zusammengefasst werden. Alle Einträge werden dabei durch ein Komma voneinander getrennt, nur die beiden letzten sollen durch das Verbindungswort “and” verbunden werden.
animals <- c("cat", "dog", "mouse", "elephant")
animals
<em>## [1] "cat" "dog" "mouse" "elephant"</em>Code-Sprache: R (r)Lösung:
Ohne das stringr Package sind zwei Aufrufe von paste() erforderlich:
- Zunächst werden alle Einträge bis auf den letzten durch ein Komma zu einem einzigen String verbunden.
- Anschließend wird das Ergebnis aus Schritt 1 mit dem letzten Vektoreintrag verknüpft.
paste(animals[-1], collapse = ", ") |> paste(animals[length(animals)], sep = " and ")
<em>## [1] "dog, mouse, elephant and elephant"</em>Code-Sprache: R (r)Das stringr package stellt mit str_flatten_comma() hierfür eine eigene Funktion mit dem sehr nützlichen last Parameter bereit:
str_flatten_comma(animals, last = " and ")
<em>## [1] "cat, dog, mouse and elephant"</em>Code-Sprache: R (r)10. arrange + distinct
Problem:
Das finale Beispiel ist durch die Arbeit an einem aktuellen Projekt von eoda inspiriert. Es liegt ein Datensatz mit zwei Spalten vor, die erste Spalte (group) beinhaltet einen Indikator für die Gruppenzugehörigkeit jeder Beobachtung. Innerhalb jeder Gruppe soll lediglich eine einzige Zeile beibehalten werden: Diejenige mit dem höchsten numerischen Wert der zweiten (value) Spalte:
set.seed(123)
data_group_value <- tibble(
group = c(1, 3, 2, 1, 1, 2, 3, 1),
value = sample(1:100, size = 8, replace = FALSE)
)
data_group_valueCode-Sprache: R (r)| group | value |
|---|---|
| 1 | 31 |
| 3 | 79 |
| 2 | 51 |
| 1 | 14 |
| 1 | 67 |
| 2 | 42 |
| 3 | 50 |
| 1 | 43 |
Lösung:
Eine mögliche Herangehensweise ist das Zusammenspiel von group_by() und slice_max():
data_group_value |>
group_by(group) |>
slice_max(value, n = 1)Code-Sprache: R (r)| group | value |
|---|---|
| 1 | 67 |
| 2 | 51 |
| 3 | 79 |
Der Nachteil hierbei ist, dass für große Datensätze gegebenenfalls sehr viele Gruppen gebildet werden, was die Effizienz der Berechnung mindert. Zudem führt dieser Ansatz bei Duplikaten nicht zu dem gewünschten Ergebnis, da slice_max() alle Beobachtungen mit dem maximalen Wert auswählt:
data_group_value_duplicates <- data_group_value |>
mutate(
value = case_when(
group == 1 ~ 20L,
TRUE ~ value
)
)
data_group_value_duplicates |>
group_by(group) |>
slice_max(value, n = 1)Code-Sprache: R (r)| group | value |
|---|---|
| 1 | 20 |
| 1 | 20 |
| 1 | 20 |
| 1 | 20 |
| 2 | 51 |
| 3 | 79 |
In diesem Fall wäre also ein zusätzlicher Aufruf von slice(1) erforderlich, um wirklich nur eine einzige Zeile pro Gruppe zu behalten.
Eine effizientere Lösung greift auf die dplyr Kombination von arrange() und distinct() zurück. Zuerst werden alle Zeilen innerhalb jeder Gruppe absteigend nach ihren value Werten sortiert. Der auszuwählende maximale Wert steht demnach stets an erster Stelle innerhalb jeder Gruppe.
Im zweiten Schritt ist ein Aufruf von distinct() ausreichend, da diese Funktion bei Duplikaten immer den zuerst auftretenden Wert beibehält und alle anderen aus der Spalte entfernt:
data_group_value_duplicates |>
arrange(group, desc(value)) |>
distinct(group, .keep_all = TRUE)Code-Sprache: R (r)| group | value |
|---|---|
| 1 | 20 |
| 2 | 51 |
| 3 | 79 |
Fazit
In diesem Beitrag haben wir die Nützlichkeit ausgewählter Tidyverse-Funktionen anhand verschiedener Beispiele veranschaulicht.
Manche Problemstellungen wären auf auch anderem Wege zu lösen
– aber nur mit größeren Aufwand
Python, R & Shiny
Unsere Trainings ebnen Ihnen den Weg für Ihre nächsten Schritte. Machine Learning, Datenvisualisierung, Zeitreihenanalysen oder Shiny:
Finden Sie bei uns den richtigen Kurs für Ihre Anforderungen.

Software:
Das Potenzial von Python & R voll ausschöpfen – mit YUNA
Mit YUNA automatisieren ganze Geschäftsprozesse und beantworten Fragen, an die Sie vorher nicht gedacht haben!

Blog:
7 hilfreiche Pandas-Funktionen, die den Tag retten können!
Noch mehr Tipps? In diesem Beitrag zeigt unser Data Scientist Matthias, welche Pandas-Funktionen, gerade am Anfang, besonders hilfreich sein können.
Training:
Erzählen Sie Ihre Data Story – interaktiv und verständlich mit Shiny
Entwickeln Sie erfolgreiche Shiny-Anwendungen – wir zeigen Ihnen wie. In unserem Shiny Training lernen Sie, wie Sie Ihre Analysen in R bestmöglich in Szene setzen.

Software:
Wir sind Ihr Posit-Partner!
Wir bieten das Komplettpaket: Von der Beratung über den Einkauf und die Integration bis zum Betrieb der professionellen Posit-Produkten in Ihrem Unternehmen – alles aus einer Hand!